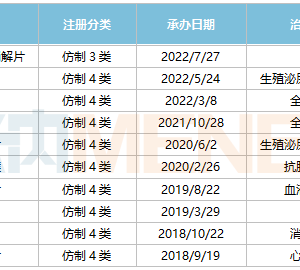
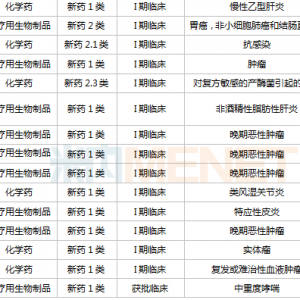
基因编辑, “何方神圣”
基因编辑是一种对靶基因或转录产物进行敲除、插入和定点突变等精确修饰的基因工程技术,主要通过人工核酸酶实现对基因组的特定基因序列的敲除、插入或精确修饰。目前,主要有三大基因编辑技术,包括:锌指核酸酶 (Zinc finger nucleases; ZFNs) 技术,转录激活因子样效应物核酸酶 (transcription activator-like (TAL) effector nucleases; TALENs) 技术和CRISPR/Cas9技术。ZFNs是最较早用于基因组编辑的人工合成的限制性内切酶,该酶是异源二聚体,包含DNA结合锌指蛋白(ZFP)结构域和非特异性FokI核酸酶结构域。DNA切割域的FokI核酸酶必须二聚化以切割DNA。该技术已被用于修饰各种生物体内的内源性基因。与ZFNs的模块化结构一样,TALENs的羧基末端也含有FokI核酸酶结构域,它借助TALEs(来源于植物致病性黄单胞菌属细菌)来识别特异性DNA碱基对。相较于ZFNs技术,其优势在于可以定点识别靶基因,从而使得基因编辑更加准确高效,其脱靶效应以及细胞毒性也得到了显著改善。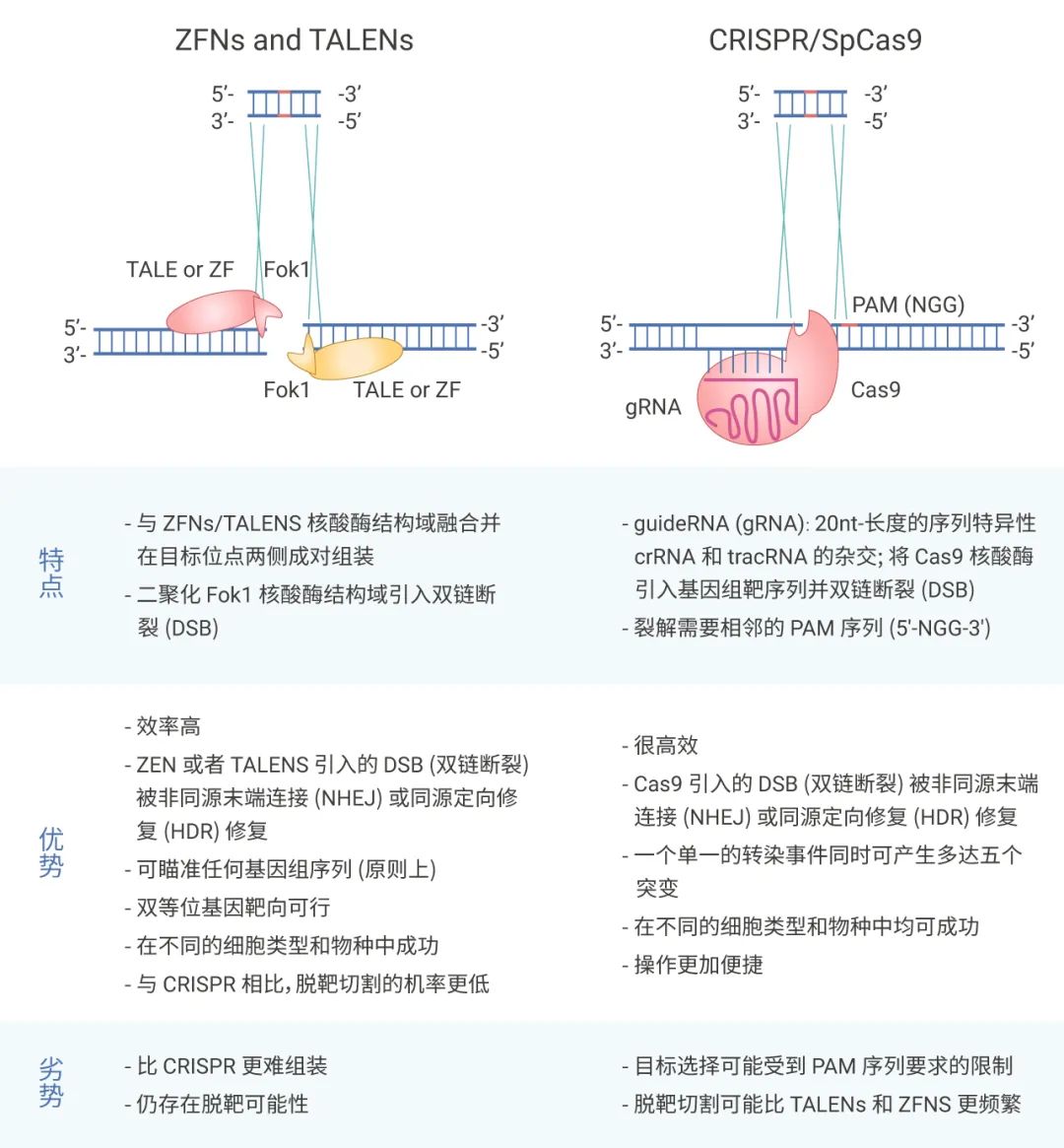
图1. 不同基因编辑手段的对比
2020年,两位女性科学家Emmanuelle Charpentier和Jennifer A. Doudna因发现CRISPR/Cas9基因剪刀,而获得2020年诺贝尔化学奖。
CRISPR/Cas系统由一小段RNA和一种高效的DNA切割酶(Cas核酸酶)组成的系统,该技术不像TALENs技术和ZFNs技术是依赖于蛋白与靶基因之间的识别,而是由sgRNA和靶基因之间形成复合物,从而完成特定基因序列的编辑。
CRISPR/Cas9技术切割效率相对较高且操作简单,并且可以同时进行多位点的编辑。总之,三种不同的基因编辑技术都有其各自的特点。今天小M的“重头戏”,CRISPR/Cas9。
CRISPR/Cas9的调控机制
在细菌及古细菌中,CRISPR系统共分成3类,其中I类和Ⅲ类需要多种CRISPR相关蛋白(Cas蛋白)共同发挥作用。而来自Streptococcus pyogenes的Ⅱ型系统只需要一种Cas蛋白(Cas9)即可发挥核酸内切酶活性。因此,CRISPR/Cas9系统应用最为广泛。CRISPR/Cas9系统已经成功应用于植物、细菌、酵母、鱼类及哺乳动物细胞。
■第一步:捕获外源DNA
Cas9蛋白包含两个核酸酶结构域:切割非互补DNA链的RuvC结构域和切割互补DNA链的HNH结构域(如图2a)。
噬菌体等外源DNA入侵时,CAS蛋白复合物通常识别3’端含NGG的前间隔序列邻近基序(PAM)区域。然后,侵入的噬菌体或质粒释放的短DNA片段(称为Protospacer),插入宿主CRISPR位点中(由重复序列隔开)。
■第二步:crRNA合成
CRISPR序列转录,形成前体CRISPRRNA(pre-crRNA)。Cas9及RNaseIII在tracrRNA与pre-crRNA上的重复序列配对形成双链RNA的条件下,对pre-crRNA进行剪切,形成成熟的tracrRNA-crRNA双链RNA(即sgRNA)。Cas9核酸酶和sgRNA形成Cas9核糖核蛋白(RNP)。
■第三步:靶向干扰
当外源DNA再次进入细胞时,Cas9蛋白携带sgRNA,去识别外源DNA的Protospacer(前间隔序列),并与之结合,通过Cas9解旋酶和核酸酶对靶基因进行剪切。造成靶基因DNA的双链断裂(DSB),从而达到干扰靶基因表达的目的,在修复断裂同时引入基因敲除或敲入。
图2. 通过非同源末端连接 (NHEJ) 或同源定向修复 (HDR) 内源性修复双链DNA断裂[2]
综上,CRISPR技术主要是利用位点特异Cas核酸酶在基因组靶位点处引入DNADSB,再经细胞自身的非同源末端连接(NHEJ)或同源重组修复(HDR)对DSB进行修复,最终实现目标基因敲除和碱基编辑等基因组遗传修饰。
CRISPR/Cas9的小分子调控策略
CRISPR技术在疾病治疗、基因功能调控、药物研发等多个方面具有广阔的应用前景,但也存在脱靶、基因毒性等副作用问题。由于Cas9蛋白和sgRNA在其自身活性、识别位点及结合能力等方面的不同特性,因此在应用中可以通过对Cas9蛋白酶以及与靶DNA的结合进行有效的调控。目前,如:遗传调节、小分子激活剂、小分子抑制剂、生物响应性输送载体以及CRISPR/Cas9系统的光/热/超声/磁激活等方法已被研究开发。
■小分子激活剂控制Cas9活性的策略
可通过添加小分子来控制Cas9的构象变化,来实现对Cas9蛋白活性的时空控制。
案例1:当内含肽插入Cas9蛋白的不同位点,Cas9核酸酶失活;添加4-HT(4-hydroxytamoxifen),通过构象变化和自切割反应去除内含肽,可重新激活Cas9蛋白(图3a)。根据内含肽的插入位点,Cas9的激活效率3倍到10倍不等,与野生型Cas9相比,这种调控策略可增加开/脱靶效应比率。
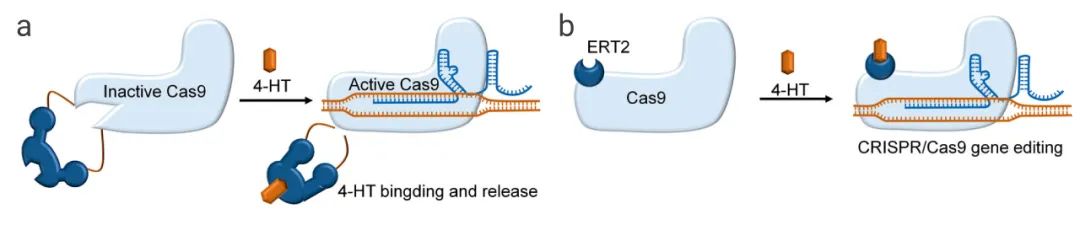
图3. 小分子激活剂控制Cas9活性的策略[9]
a:通过4-HT结合,恢复失活的Cas9活性;b:Cas9和雌激素受体(ERT2)的融合被分离在细胞质中,通过添加4-HT使得融合物进入细胞核,形成Cas9/sgRNA复合物
案例2:基于化学诱导的Cas9蛋白分裂片段的二聚化。Zetsche等人设计了不同的分裂位点(Arg535和Glu573)生成分裂Cas9(split-Cas9)蛋白,同时产生C端和N端Cas9片段(可分别与FK506结合蛋白(FKBP)和FKBP雷帕霉素结合结构域(FRB)结合)。这种方法通过雷帕霉素诱导的异源二聚作用实现了split-Cas9的条件重构和激活。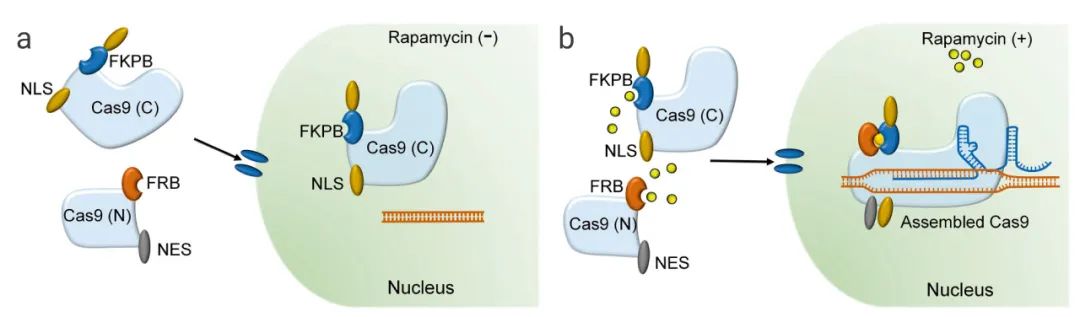
图4.小分子激活剂控制Cas9活性的策略[9]
■小分子抑制剂控制Cas9活性的策略
由于Cas9活性的升高和持续可能会导致脱靶效应、染色体易位和遗传毒性,因此在目标编辑后,Cas9核酸酶活性必须迅速限制在一个狭窄的时间范围内。
如下图5所示,DHFR(ER50)是一个不稳定的结构域,可快速靶向Cas9蛋白进行蛋白酶体介导的Cas9降解,但添加小分子抑制剂甲氧苄氨嘧啶(TMP)或4-OHT后可以使其稳定。用Cas9-DHFR或Cas9-ER50系统编辑VEGFA基因时,用不同剂量的TMP或4OHT剂量依赖性控制靶向VEGFA基因的复合物Cas9-DHFR(ERR50)的靶向和非靶向活性。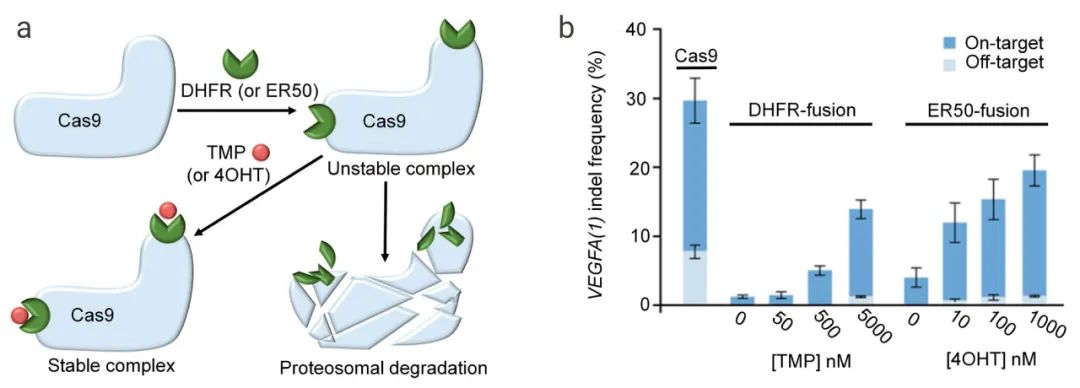
图5.小分子抑制剂控制Cas9活性的策略[9]
总结
CRISPR-Cas9系统已成为一种在任何细胞基因组的精确位置进行修改的有效工具。Cas9介导的DNA切割后发生的主要细胞修复途径是错误的非同源末端连接(NHEJ)途径。同源定向重组(HDR)的效率远远低于NHEJ。因此,降低NHEJ的频率,同时增加Cas9介导的DNA切割后的HDR效率,从而提高基因组编辑的整体效率和精准度成为科学家关注的焦点。与此同时,通过研究人员的不断努力,生物活性小分子提供了一种简单而有效的调控策略,可进一步拓宽精确基因组编辑的应用范围。
参考文献
1. Gibson GJ, Yang M.Whatrheumatologists need to know about CRISPR/Cas9. Nat Rev Rheumatol. 2017 Apr;13(4):205-216.
2. Westermann L, Neubauer B, Köttgen M. Nobel Prize 2020 in Chemistry honors CRISPR: a tool for rewriting the code of life. Pflugers Arch. 2021 Jan;473(1):1-2.
3. Dominguez AA, Lim WA, Qi LS. Beyond editing: repurposing CRISPR-Cas9 for precision genome regulation and interrogation. Nat Rev Mol Cell Biol. 2016 Jan;17(1):5-15.
4. Matano M, Date S, Shimokawa M, Takano A, Fujii M, Ohta Y, Watanabe T, Kanai T, Sato T. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids. Nat Med. 2015 Mar;21(3):256-62.
5. Yang H, Wang H, Shivalila CS, Cheng AW, Shi L, Jaenisch R. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 2013 Sep 12;154(6):1370-9.
6. Sánchez-Rivera FJ, Jacks T. Applications of the CRISPR-Cas9 system in cancer biology. Nat Rev Cancer. 2015 Jul;15(7):387-95.
7. Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 2012 Aug 17;337(6096):816-21.
8. Zalatan JG, Lee ME, Almeida R, Gilbert LA, Whitehead EH, La Russa M, Tsai JC, Weissman JS, Dueber JE, Qi LS, Lim WA. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 2015 Jan 15;160(1-2):339-50.
9. Zhuo C, Zhang J, Lee JH, Jiao J, Cheng D, Liu L, Kim HW, Tao Y, Li M. Spatiotemporal control of CRISPR/Cas9 gene editing. Signal Transduct Target Ther. 2021 Jun 20;6(1):238.
10. Maji B, Gangopadhyay SA, Lee M, Shi M, Wu P, Heler R, Mok B, Lim D, Siriwardena SU, Paul B, Dančík V, Vetere A, Mesleh MF, Marraffini LA, Liu DR, Clemons PA, Wagner BK, Choudhary A. A High-Throughput Platform to Identify Small-Molecule Inhibitors of CRISPR-Cas9. Cell. 2019 May 2;177(4):1067-1079.
本网页由机器采集生成,若侵权请及时联系删除。
原文链接:https://med.sina.com/article_detail_103_2_114064.html